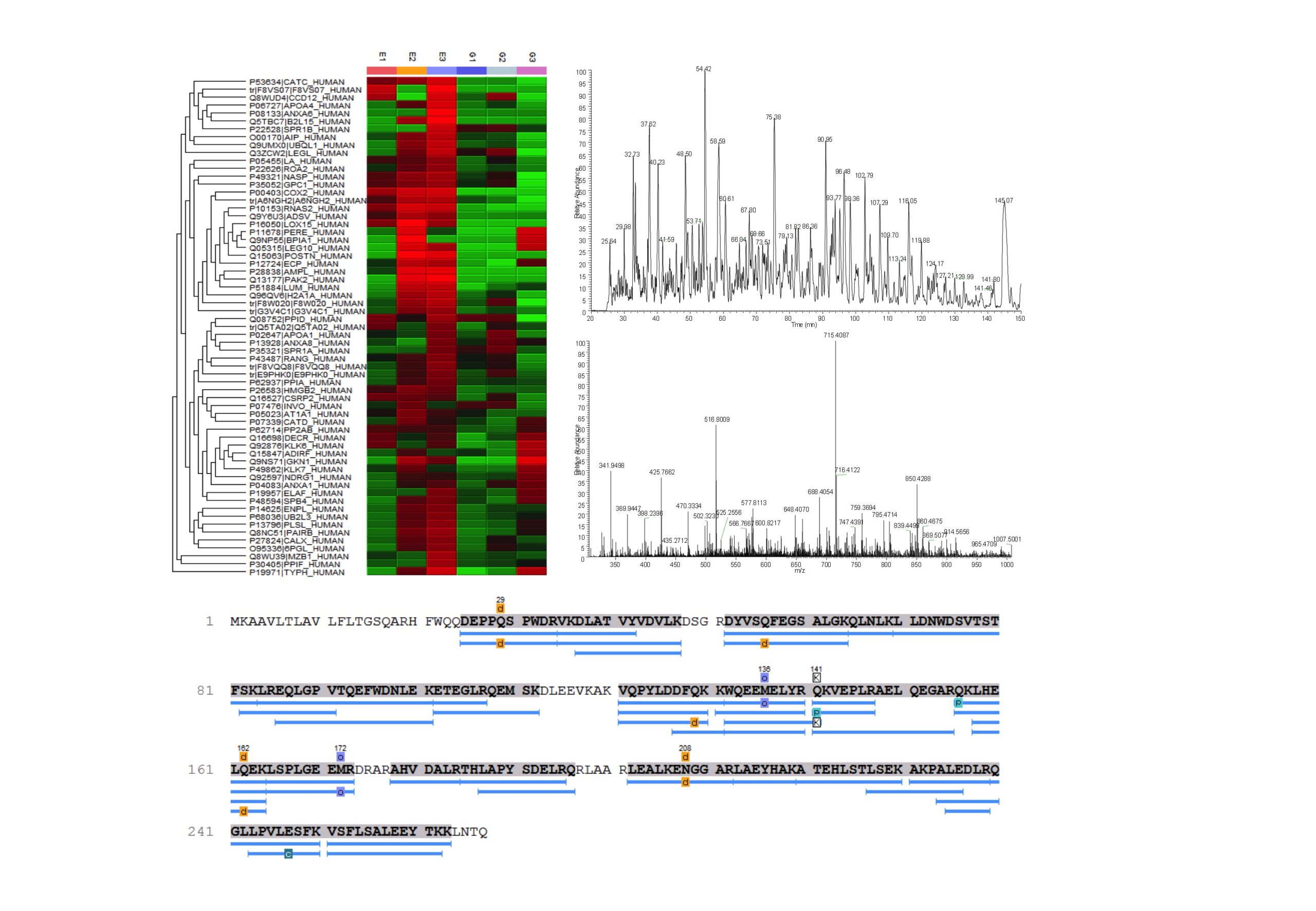
Proteomik von Pollen-Allergenen


Nachdem wir uns viele Jahre mit Biochemie und Molekularbiologie der Hefesporenwand befasst hatten, konzentriert sich unsere Gruppe mittlerweile auf Massenspektrometrie und Proteomik von Allergenen.
Mit der Entwicklung moderner Tandem-Massenspektrometer wurde die Proteomforschung zu einem immer wichtigeren Instrument zur Identifizierung und Charakterisierung von Proteinen in komplexen Proben.
Grundsätzlich sind zwei komplementäre Ansätze möglich:
- Proteine werden aus Zellen oder Organellen extrahiert, mit Protease verdaut und die resultierenden Peptide werden durch Umkehrphasen-HPLC getrennt und durch ein direkt gekoppeltes Tandem-Massenspektrometer analysiert.
- Die andere Möglichkeit besteht darin, die extrahierten Proteine durch ein- oder zweidimensionale Gelelektrophorese zu trennen, Proteinspots mit Protease zu verdauen und die Peptide durch MS zu analysieren.
Tandem-MS-Instrumente können gleichzeitig Peptidmassen und Peptidfragmentierungsmuster erfassen. Mit diesen Informationen können Proteine in Proteindatenbanken eindeutig identifiziert werden. Sequenzinformationen, die für unbekannte Proteine erhalten wurden, können zum Klonieren der entsprechenden Gene verwendet werden.
Unsere Gruppe verwendet ein Q Exactive Orbitrap-Massenspektrometer mit Nano-Elektrospray-Ionisierung, optional gekoppelt mit einer Kapillar-HPLC. Mit der oben beschriebenen Methode versuchen wir, neue Proteine oder Proteinisoformen zu identifizieren, die bei allergischen Patienten Typ-I-Allergien auslösen.
Unser Hauptaugenmerk liegt auf Allergenen aus Baumpollen (Birken und ausgewählte Bäume) und Unkrautpollen (Beifuß, Wolfsmilch). Unsere Arbeit erfolgt in enger Zusammenarbeit mit der Arbeitsgruppe von Fátima Ferreira.